当前位置 :首页>新闻动态
5月2日,南方科技大学深港微电子学院李毅副教授课题组在DNA信息存储与隐私通信领域取得重要进展,在国际顶尖学术期刊《自然·通讯》(Nature Communications)上发表了题为 “De novo non-canonical nanopore basecalling enables private communication using heavily-modified DNA data at single-molecule level”的研究论文。(https://www.nature.com/articles/s41467-025-59357-2)
随着信息技术飞速发展,数据安全与隐私保护面临日益严峻的挑战。DNA作为一种极具潜力的信息存储介质,因其高密度、长寿命的特点备受关注。然而,如何利用DNA进行安全的信息传输,防止信息被轻易窃取,是一个关键科学问题。传统的DNA测序技术和碱基识别器(Basecaller)主要针对天然DNA设计,对于经过化学修饰的DNA(可用于隐藏信息)往往无法准确读取,这为基于DNA的隐私通信提供了思路,但也带来了信息解密的巨大挑战,阻碍了其应用。
针对这一挑战,李毅课题组创新性地提出了一种基于纳米孔测序和深度学习的框架——DeepSME。该框架能够对经过重度化学修饰(例如,使用5-羟甲基胞嘧啶(5hmC)替换所有天然胞嘧啶C碱基)的DNA进行“从头构建”(de novo)碱基识别器。这种非天然的化学修饰会严重干扰传统碱基识别器的判读,导致信息读取失败,从而有效“隐藏”信息,保护通信隐私。而DeepSME则如同专门匹配的“密钥”,能够精准解密这些被隐藏的分子信息,实现DNA层面的高私密性、高安全性通信。
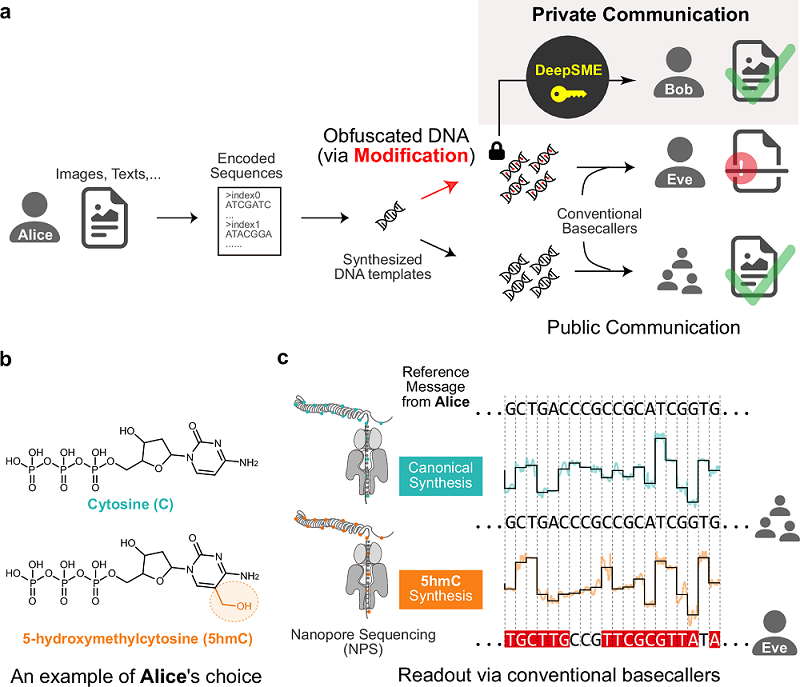
图1:基于化学修饰DNA和DeepSME的私密通信示意图
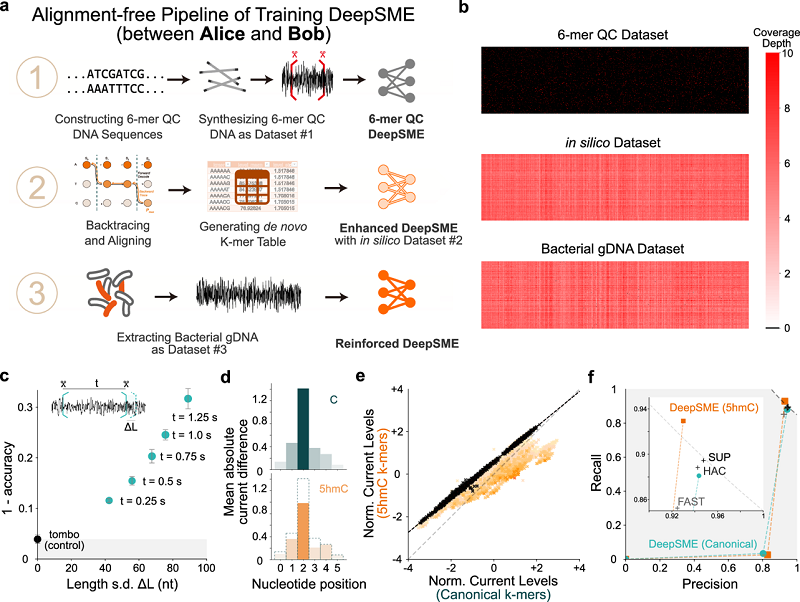
图2:DeepSME训练流程概览及其性能评估
研究团队开发了一种创新的三阶段、无需精确序列比对(alignment-free)的训练流程来构建DeepSME(图2a-b)。该方法从零开始生成k-mer(DNA短序列片段)字典,克服了修饰DNA缺乏对齐手段和现有模型的难题。最终训练得到的DeepSME碱基识别器在精度(Precision)和召回率(Recall)上均超过92%(图2f),解码综合性能指标(F1分数)达到86.4%,显著优于当前所有最优的商业或开源碱基识别器(图3f)。
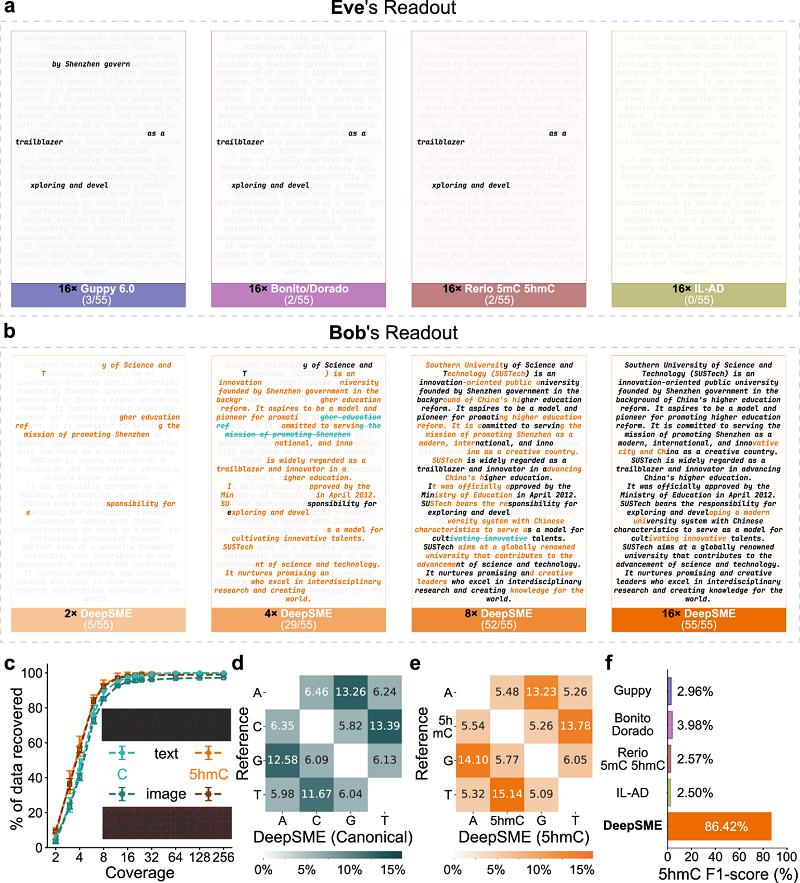
图3:DeepSME成功解密隐藏在修饰DNA中的文本信息
实验结果表明,配合本课题组开发复合对冲DNA编码方案(Composite Hedges Nanopores,https://www.nature.com/articles/s41467-024-53455-3)的DeepSME能够成功解密隐藏在完全5hmC修饰DNA中的文本和图像文件。如图3所示,对于不知情的第三方(Eve),使用现有的商业或开源碱基识别器几乎无法恢复任何有效信息。而对于拥有DeepSME“密钥”的接收方(Bob),在仅需16倍测序深度下,即可高效并准确地恢复全部原始文本信息(图3b)。这一结果有力证明了DeepSME框架在DNA私密通信中的可行性和优越性。
该研究不仅为DNA数据存储和传输提供了一种新颖、高效的隐私保护方案,也展示了深度学习在解读复杂生物分子信号方面的强大潜力。DeepSME框架具有无需比对、可定制、训练高效、计算资源要求相对适中等优点,未来有望应用于更广泛的生物工程、信息安全、防伪溯源及精准医疗领域。
南方科技大学为论文第一单位,深港微电子学院硕士研究生樊青远为第一作者,深港微电子学院李毅副教授为唯一通讯作者。该研究工作得到了国家重点研发计划、国家自然科学基金、广东省基础与应用基础研究基金、深圳市科技创新委员会基础研究项目以及南方科技大学科学与工程计算中心等单位的支持。